2019年12月6日,复旦大学类脑智能科学与技术研究院贾天野青年研究员与英国伦敦国王学院Sylvane Desrivières副教授合作在《分子精神病学》(Molecular Psychiatry)在线发表题为《皮质下脑体积与血液中全表观遗传组关联性的元分析:来自ENIGMA表观遗传工作组的发现》(“Epigenome-wide meta-analysis of blood DNA methylation and its association with subcortical volumes: findings from the ENIGMA Epigenetics Working Group”)的研究论文。该项研究通过与人类行为/精神障碍密切相关的神经影像的表观遗传学位点,发现血液及大脑中的DNA甲基化对大脑皮质下区域体积的影响。
在表观遗传学中,DNA甲基化是一种重要的基因修饰机能,能够显著地影响被修饰基因的表达,进而影响受到此基因调控的性状。和具有高度稳定性的基因不同,DNA甲基化会随着年龄和外部环境的影响而改变,因而研究DNA甲基化可以一定程度的反应环境基因交互作用。此外,相比在组织间特异性极高的基因表达而言,DNA甲基化在不同组织和器官之间具有较高的稳定性,因此研究血液中DNA甲基化对性状的影响可以很大程度上类比于大脑中DNA甲基化的效果。
贾天野青年研究员与Sylvane Desrivières副教授一起组织国际脑影像遗传学合作研究计划ENIGMA中来自全世界11个甲基化工作组的数据,在3337名被试中,发现了与人类行为/精神障碍密切相关的神经影像的表观遗传学位点,并原创性地提出了大脑-血液中介分析原理,不仅可用于确认血液中的表观遗传学标记物与大脑中对应标记物的等效性,并可推广用于回答表观遗传学及表达组学分析中广泛存在的不同器官/组织间标记物的等效性问题。
研究团队首先通过全表观遗传组关联分析(EWAS)的荟萃分析(meta-analysis)发现了cg26927218(BAIAP2)和cg17858098(ECH1)两个DNA甲基化位点与海马体的体积相关(图1)。
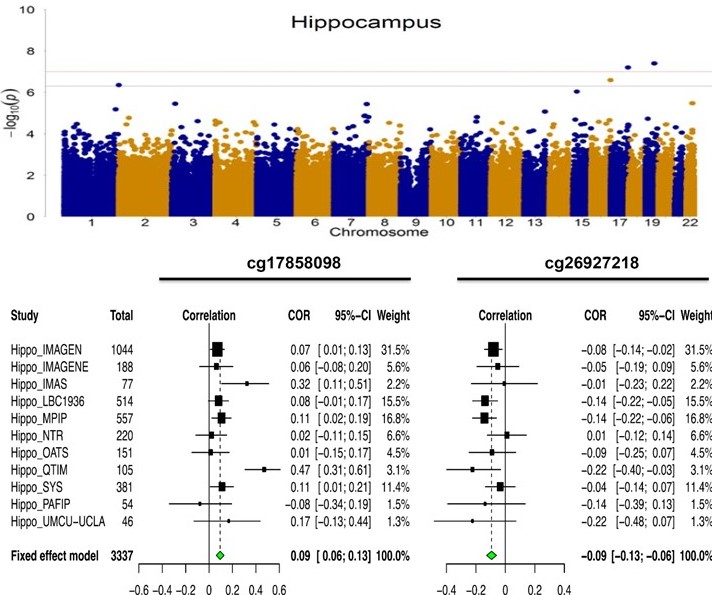
图1:大脑海马体体积的全表观遗传组关联分析结果
此外,基于DNA甲基化的聚簇现象(即CpG islands),使用聚合分析(cluster-level analysis)comb-P方法再次分析上面得到的EWAS的结果,并在全基因组上寻找到了与海马体体积相关的20个聚集的甲基化差异区域(differentially methylated region, DMR)。其中,cg26927218(BAIAP2)再次被找到,在此基础上,另有三个DMR(分别位于CMYA5,HHEX和CPT1B中)还可以在至少2个不同的子群体中通过comb-P发现显著。
接下来,研究团队在独立数据库中确认了这些DMR在大脑中对相关基因表达的影响;更进一步的,建立了一套全新的中介统计模型来确认血液与大脑中表观遗传学标记物的等效性,即通过比较独立数据库中大脑-血液DNA甲基化在DMR1-3中所有甲基化位点上各自的相关程度(个体维度相关)与荟萃分析结果中同样的每个DNA甲基化位点和海马体积的相关程度(个体维度相关)的相似度(甲基化位点维度相关),发现血液DNA甲基化与海马体积的相关是经由大脑(上颞叶)的DNA甲基化程度中介(r=0.751,矫正脑区数量后P=0.0066),从而证明了血液和大脑中DNA甲基化对大脑体积影响的等效性(图2)。事实上,这一思路为表观遗传学(以及表达组学)中检验生物学标记物在不同器官/组织间的等效性问题提供了一套普适且高效的验证方案。

图2:血液-大脑DNA甲基化与海马体体积的中介分析结果
最后,研究团队发现EWAS得到的备选基因(BAIAP2,CMYA5,HHEX和CPT1B)的已知功能都和海马体的功能直接相关(例如记忆和老年痴呆症),再一次验证了我们找到的甲基化位点在医学及相关科研上的重要意义。
贾天野青年研究员为该论文的第一作者,Sylvane Desrivières副教授为通讯作者。该研究得到了国家自然科学基金委、上海市浦江人才计划、上海市脑与类脑智能基础转化应用研究市级重大专项以及上海市科委科技创新计划等的经费支持。